Capítulo 3 R para ciencia de datos
Emilio L. Cano
Universidad Rey Juan Carlos
3.1 Introducción
El análisis estadístico de datos es una tarea fundamental en la transformación digital de las empresas y organizaciones. Siempre ha estado ahí, pero en la actualidad la disponibilidad de datos, la cantidad de los mismos y la velocidad con la que se requieren resultados están haciendo necesario el capacitar a los profesionales para su análisis con nuevas herramientas. Nuevas tendencias (muchas veces malinterpretadas), como Inteligencia Artificial (IA), Big Data, Industria 4.0, Internet of Things (IoT) o Data Science, aumentan el interés por parte de las empresas, los profesionales y los investigadores en estas técnicas.
El tratamiento de datos y su análisis requiere el uso de software avanzado. Aunque algunas tareas como, por ejemplo, mecanizar y almacenar datos, se pueden realizar eficazmente con programas de hoja de cálculo como Excel, se debería utilizar software especializado para el análisis de datos. Existen distintos paquetes estadísticos comerciales, como SPSS, Statgraphics, Stata, SAS, JMP o Minitab. En los últimos años se ha abierto camino como alternativa el software estadístico y lenguaje de programación R (R Core Team, 2023). Hay otras alternativas que, en su mayoría, o son parciales, referidas a un ámbito concreto, o son más lenguajes de programación que software estadístico, como Python. R es software libre, pero su gratuidad solo es una de sus ventajas, como se verá a lo largo del libro. Su gran inconveniente es la curva de aprendizaje: no es tan fácil de aprender y usar como un software de ventanas, ya que el uso de R se basa en expresiones que hay que ejecutar desde scripts (archivos de código).
R es un sistema para computación estadística: software de análisis de datos y lenguaje de programación. Ha sido ampliamente utilizado en investigación y docencia, y actualmente también en las empresas y organismos públicos. Es la evolución del trabajo de los laboratorios Bell con el lenguaje S (Venables & Ripley, 2002), llevado al mundo del software libre por Ross Ihaka y Robert Gentleman en los años 90 (Ihaka & Gentleman, 1996). La version R 1.0.0 se publicó el 29 de febrero de 2000.
Uno de los aspectos más espectaculares de R es la cantidad de paquetes disponibles. Un paquete (package) de R es un componente con funcionalidad adicional que se puede instalar en el sistema para ser utilizado por R. En el momento de compilar este libro, el número de paquetes disponibles en el repositorio oficial es de 20250.
Una vez conocido el mundo de R, se plantea la siguiente pregunta: ¿y por qué utilizar R? Es imposible dar un único motivo. A continuación se enumeran algunos de ellos:
- Es Free and Open Source Software (FOSS). Gratis y libre. En inglés se suele decir free as in free beer, and free as in free speech.
- Tiene una amplia comunidad de usuarios que proporciona recursos.
- Es multiplataforma.
- Se usa cada vez en más empresas e instituciones.
- Es posible obtener soporte comercial, por ejemplo a través de Posit Software PBC.3
- Se ha alcanzado una masa crítica de usuarios que lo hace confiable.
- Es extensible (desde pequeñas funciones, hasta paquetes).
- Se puede implementar la innovación inmediatamente. En software comercial hay que esperar a nuevas versiones, en el mejor de los casos.
- Posee características de “investigación reproducible”. En el Cap. 44 se tratará qué implica este enfoque. En contextos distintos a la investigación, se puede hablar de informes reproducibles y trazabilidad del análisis.
Por otra parte, el uso de R en las empresas está creciendo exponencialmente debido, principalmente, a la necesidad de analizar y visualizar datos con herramientas potentes para explotar todo su potencial. Grandes empresas de todos los sectores llevan tiempo utilizándolo, si bien la popularización del software y su conocimiento entre los nuevos titulados está facilitando que empresas de todo tipo y tamaño aprovechen esta herramienta en su estrategia digital. Así, además de la visualización y presentación efectiva de los datos, equipos bien formados pueden descubrir relaciones entre variables clave, realizar predicciones, tomar mejores decisiones o mejorar sus procesos gracias al análisis avanzado de datos más allá de la hoja de cálculo.
3.2 La sesión de R
R es una aplicación de análisis estadístico y representación gráfica de datos, y además un lenguaje de programación. R es interactivo, en el sentido de que responde a través de un “intérprete” a las entradas que recibe a través de la consola.
La interfaz de usuario de R (R GUI, Graphical User Interface) cumple las funciones básicas para interactuar con R, pero es muy pobre a la hora de trabajar con ella. En su lugar, es más conveniente utilizar el entorno de desarrollo RStudio Desktop (o su versión en la nube https://posit.cloud/), que es como un “envoltorio” del sistema R con más funcionalidades y ayudas, pero manteniendo el mismo nivel de interacción: consola y scripts.4 Al igual que R, RStudio es una aplicación de software libre, pero, en este caso, desarrollada y mantenida por la compañía privada Posit PBC.
Nota
Una cosa muy importante en R es que las expresiones son sensibles a mayúsculas,
y por tanto los objetos datos y Datos son distintos.
3.3 Instalación de R
Durante todo el libro se utiliza la interfaz RStudio. Pero, como se avanzó anteriormente, RStudio es solo un “envoltorio” de R, por lo que previamente hay que tener instalado en el ordenador el sistema “base” de R. R está disponible para sistemas Windows, MacOS y Linux. Por cuestiones de espacio, no se incluyen detalles en este libro, pero la instalación es sencilla siguiendo las instrucciones en sus correspondientes websites:
- Instalación de R: http://www.r-project.org
- Instalación de RStudio: https://posit.co
Para completar la instalación de R, se muestra cómo instalar5 los paquetes del tidyverse6 mediante expresiones en la consola o script con la función install.packages():
install.packages(pkgs = "tidyverse")Una vez instalado el paquete, se cargará con la instrucción library("nombre_paquete") en la sesión de R donde se quiera utilizar.
A veces resulta útil usar directamente
la función que se va a utilizar en vez de cargar todo el paquete. Esto se hace con el operador ::, es decir, nombre_paquete::funcion(). La siguiente expresión serviría para usar
la función select() del paquete dplyr sin cargar el paquete entero.
dplyr::select()3.4 Trabajar con proyectos de RStudio
La manera más eficiente de trabajar con R es mediante proyectos de RStudio. Esto permite abstraerse de los detalles de la sesión de R (espacio de trabajo, directorio de trabajo, environment), ya que al abrir un proyecto estará todo preparado para seguir el trabajo donde se dejó, o empezar de cero si se acaba de crear. Para crear un proyecto de RStudio, se despliega el menú de proyectos a la derecha en la barra de herramientas y se selecciona “New Project…” También se puede hacer en el menú “File/New Project…”.
Es aconsejable crear siempre una estructura de carpetas que permita tener todo organizado desde el principio, porque al final los proyectos crecen. La estructura perfecta no existe, y depende del proyecto particular. Las siguientes carpetas pueden ser útiles en un amplio abanico de proyectos, y las tres primeras se pueden usar prácticamente en cualquier proyecto:
-
data: en esta carpeta se tienen los archivos de datos, tanto aquellos orígenes de datos que se quieran importar como los que se puedan guardar desde un script. -
R: para los scripts. Es posible que solamente haya un script en nuestro proyecto, pero si hubiera más se pueden guardar en esta carpeta. -
inform: aquí se pueden guardar los archivos Quarto o R Markdown que se utilicen para generar informes o presentaciones. -
img: si en nuestro proyecto se utilizan imágenes de cualquier tipo, es una buena idea tenerlas en una carpeta aparte. -
test: si se quieren separar los scripts que se utilicen para pruebas y no se quieren mezclar con los “buenos” en la carpetaR. -
aux,tmp,util,notas,doc, …: este tipo de carpetas vienen bien cuando hay información que está relacionada o es útil para un proyecto, pero el archivo no es del proyecto de análisis de datos en sí. Por ejemplo, unas especificaciones de un producto o servicio, un artículo científico, fotografías de una fábrica, comunicaciones con clientes, etc. -
ejercicios,practicas, …: si nuestro proyecto forma parte de una asignatura, curso o similar.
Nota
Un aspecto importante cuando se trabaja en proyectos colaborativos es el control de versiones. Este tema se aborda en el Cap. 46.
3.5 Tratamiento de datos con R
En este apartado se van a empezar a utilizar expresiones de R. Las expresiones se escribirán en scripts, que pueden contener “comentarios” (texto que no se ejecutará) utilizando el símbolo “almohadilla” (#). Muchas de las expresiones que se usan son llamadas a funciones.7 La ayuda de cualquier función se puede obtener en la consola usando la expresión ?function, donde function es el nombre de la función u objeto del que se quiere obtener ayuda.
3.5.1 Estructuras y tipos de datos
Las estructuras y tipos de datos más frecuentes con los que se trabaja en R son los que se detallan a continuación.
Tablas de datos. Son colecciones de variables numéricas y/o atributos
organizadas en columnas,
en las que cada fila se corresponde con algún elemento en el que se han
observado las características que representan las variables. La forma más común
es el data.frame. Cada columna del data.frame es, en realidad, otra estructura de datos, en concreto, un vector. Un ejemplo de data.frame es el conjunto de datos tempmin_data del paquete CDR que se analiza en el Cap. 40 y del que se muestran a continuación las primeras tres filas con la función head().
library("CDR")
head(tempmin_data, 3)
#> fecha indicativo tmin longitud latitud
#> 1 2021-01-06 4358X -4.7 -5.880556 38.95556
#> 2 2021-01-06 4220X -7.0 -4.616389 39.08861
#> 3 2021-01-06 6106X 4.7 -4.748333 37.02944Un data.frame es un objeto de datos en dos dimensiones, en el que las filas
son la dimensión 1 y las columnas la dimensión 2. Los datos se pueden “extraer”
de un data.frame por filas, por columnas o por celdas. Para extraer una
de las variables del data.frame se suele utilizar el operador $ después del
nombre del data.frame, y a continuación el nombre de la variable.
El operador <- asigna al “símbolo” que hay a su izquierda el resultado
de la expresión que hay a su derecha, y lo guarda con ese nombre
en el espacio de trabajo.8 Por ejemplo, la siguiente expresión extrae todas las filas de la columna tmin o, dicho de otra forma, el vector con todas las temperaturas mínimas registradas y lo guarda en el objeto temp_min.
temp_min <- tempmin_data$tmin
Vectores y matrices. Ya se ha visto que una columna de una tabla de datos es un vector. También se pueden crear vectores con la función c() y los elementos del vector separados por comas. Una matriz es un vector organizado en filas y columnas. A modo de ejemplo, la primera de las siguientes expresiones crea un vector llamado nombres con dos cadenas de texto, y la segunda crea una matriz numérica llamada coordenadas a partir de las columnas 4 y 5 del conjunto de datos tempmin_data. Nótese que la extracción de valores de un conjunto de datos o de una matriz se puede realizar también por sus índices de filas y columnas entre corchetes separados por una coma. En este caso se extraen todas las filas (pues no se especifica ninguna en la dimensión 1) de las columnas 4 y 5.
Factor. Es un tipo especial de vector para representar variables categóricas (también denominadas atributos o factores). En general, una variable categórica suele tomar un número reducido
de valores diferentes (categorías), identificados con etiquetas (labels) y que se llaman
niveles del factor (levels).
Un ejemplo es el dataset dp_entr del paquete CDR que se analiza en el Cap. 24. La columna ind_pro11 es un indicador que toma los valores S y N, mientras que des_nivel_edu toma tres posibles valores.
dp_entr[1:5, c(1, 17)]
#> ind_pro11 des_nivel_edu
#> 1 S MEDIO
#> 497 N MEDIO
#> 265 N BASICO
#> 534 N MEDIO
#> 415 N BASICO
levels(dp_entr$des_nivel_edu)
#> [1] "ALTO" "BASICO" "MEDIO"Listas. Son estructuras de datos que contienen una colección de elementos indexados que, además, pueden tener un nombre. Pueden ser heterogéneas, en el sentido de que cada elemento de la lista puede ser de cualquier tipo.
A modo de ejemplo, se muestran los nombres del objeto tempmax_data del paquete CDR, que contiene seis elementos de distintas clases.
names(tempmax_data)
#> [1] "ESP" "ESP_utm" "grd_sf" "grd_sp"
#> [5] "temp_max_utm_sf" "temp_max_utm_sp"
Fechas. Son un tipo de datos especial que algunas veces provoca problemas
al compartir datos entre programas. El conjunto de datos tempmin_data contiene la columna fecha, que puede convertirse de manera inmediata a tipo fecha (Date) porque viene en un formato estándar (véase la ayuda de strptime para especificar otros formatos). El paquete lubridate del tidyverse contiene funciones para hacer más fácil el trabajo con fechas.
Cadenas de texto. Son estructuras de datos que aparecen en forma de vector de caracteres. La columna indicativo del conjunto de datos tempmin_data es un ejemplo de este tipo de datos. La ayuda de ?regexpr proporciona la información necesaria sobre cómo extraer texto con expresiones regulares, y la de ?paste, para aprender a unir cadenas de texto. El paquete stringr del tidyverse contiene funciones para facilitar el trabajo con cadenas de texto.
head(tempmin_data$indicativo)
#> [1] "4358X" "4220X" "6106X" "9698U" "4410X" "1331A"3.5.2 Importación de datos
En el apartado anterior se han utilizado tablas de datos que están incluidas en un paquete de R. Pero lo habitual es que los datos se tengan que importar de fuentes externas, como ficheros. A continuación, se describen algunas de las formas de importar los tipos de ficheros más habituales.9
Excel. Sin duda una forma muy popular de organizar los datos en ficheros es
mediante hojas de cálculo como Microsoft Excel.
Hay varios paquetes con los que se puede trabajar con archivos de Excel. En este libro se utiliza
el paquete readxl del tidyverse. Con la siguiente expresión se puede descargar un archivo Excel de ejemplo.10
download.file(url = "http://emilio.lcano.com/b/adr/p/datos/RRHH.xlsx",
destfile = "data/RRHH.xlsx",
mode = "wb")Una vez el archivo está en el directorio de trabajo de la sesión de R, se puede importar su contenido al espacio de trabajo con la siguiente expresión:
rrhh <- readxl::read_excel("data/RRHH.xlsx")Texto. Los archivos de texto son el formato más utilizado y conveniente para compartir datos. Es también
muy común que el equipamiento o el software genere datos en formato de texto. Estos
archivos suelen tener extensión .csv (comma separated values) o .txt, aunque
pueden tener cualquier otra, o incluso no tener extensión. A modo de ejemplo, con la siguiente expresión se puede descargar un archivo csv.
download.file(url = "http://emilio.lcano.com/b/adr/p/datos/ejDatos.csv",
destfile = "data/ejDatos.csv")Si el archivo tiene extensión .csv, como el anterior, vendrá
ya con una especificación muy concreta, pudiéndose usar directamente las funciones
read.csv() o read.csv2() para tener la tabla de datos en el espacio de trabajo.
merma <- read.csv2("data/ejDatos.csv")La función genérica de R para importar datos de texto es read.table(), que puede importar cualquier especificación cambiando los argumentos adecuados.
Por ejemplo, la siguiente expresión tendría el mismo resultado que se ha
obtenido con la función read.csv2:11
merma <- read.table(file = "data/ejDatos.csv",
header = TRUE,
sep = ";",
dec = ",",
fileEncoding = "utf-8")Nota
Para saber cómo importar datos desde sistemas gestores de bases de datos véase el Cap. 5.
Hay infinidad de otras fuentes de las que se pueden importar datos a R. Por ejemplo, el paquete rvest, que forma parte
del tidyverse, se puede utilizar para obtener datos de páginas web y otras fuentes de Internet, lo que se suele llamar web scraping. Por ejemplo, supóngase que se quiere importar la tabla con los datos de comunidades y ciudades autónomas españolas del enlace https://www.ine.es/daco/daco42/codmun/cod_ccaa_provincia.htm.
Las siguientes expresiones importan esta tabla al conjunto de datos ccaa_ine.
library("rvest")
url <- "https://www.ine.es/daco/daco42/codmun/cod_ccaa_provincia.htm"
ccaa_ine <- url |>
read_html() |>
html_node(xpath = '//*[@id="contieneHtml"]/table') |>
html_table(fill = TRUE)La ruta o “xpath” se puede obtener usando las herramientas de desarrollo del navegador, y puede que una vez importada la tabla se requiera algún postprocesamiento antes de poder analizar los datos.
3.5.3 Exportación de datos y archivos de datos específicos de R
En algunos proyectos es necesario guardar ciertos datos que se han ido creando o
transformando, bien para compartir con otras partes interesadas, bien para
ser utilizados en el mismo u otros proyectos.
Para exportar los datos a Excel, se utiliza la función write.xlsx() del paquete openxlsx (si no está instalado, se instala de la forma habitual).
Si lo que se quiere es exportarlo a texto, se pueden utilizar los equivalentes
a las funciones de importación write.csv(), write.csv2() o write.table().
La siguiente expresión exporta la tabla de datos tempmin_data a ficheros Excel y csv (formato en inglés).
openxlsx::write.xlsx(x = tempmin_data,
file = "data/temp_min_Filomena.csv")
write.csv(x = tempmin_data, file = "data/temp_min_Filomena.csv")También se pueden guardar los datos en formato “nativo” de R. Los archivos .RData almacenan un espacio de trabajo entero, y por tanto
pueden guardar varios objetos en el mismo archivo. Cuando posteriormente se importe,
los objetos estarán en el espacio de trabajo con su nombre original.
Se guardan con la función save() y se restauran con la función load(), como en el siguiente ejemplo.
save(tempmin_data, tempmax_data,
file = "data/datos_temperaturas.RData")
load("data/datos_temperaturas.RData") #carga de nuevo el objetoLos archivos .rds almacenan un único objeto en un archivo. Cuando posteriormente se quieran importar,
hay que asignar el resultado al nombre que se quiera. Se guardan con la
función writeRDS() y se restauran con la función readRDS(), como en el siguiente ejemplo.
saveRDS(object = tempmin_data,
file = "data/datos_temperaturas.rds")
nuevo_objeto <- readRDS(file = "data/datos_temperaturas.rds")El paquete foreign de R base y otros paquetes especializados pueden
exportar datos a otros formatos de archivo, que no se tratan
en detalle en este capítulo.
3.6 Organización de datos con el tidyverse
3.6.1 El tidyverse y su flujo de trabajo
El tidyverse es, según se define en su propia página web,12 un conjunto de paquetes de R “opinables” diseñados para ciencia de datos. Las principales ventajas (opinables) de utilizar el tidyverse son tres:
- Utiliza una gramática, estructuras de datos y filosofía de diseño común.
- El flujo de trabajo es más fluido y, una vez se comprenden las ideas principales, más intuitivo.
- Para la mayoría de las operaciones, es computacionalmente más eficiente.
Uno de los paquetes más populares del tidyverse es ggplot2, que proporciona una “gramática de gráficos”
(Wickham, 2016) y es una pieza clave del tidyverse actual, junto con los paquetes dplyr (gramática para la manipulación de datos) y tidyr (herramienta para crear datos tidy).
El flujo de trabajo propuesto por el tidyverse se describe en el libro
R for Data Science (Wickham & Grolemund, 2016) y se sintetiza en la Fig. 3.1.
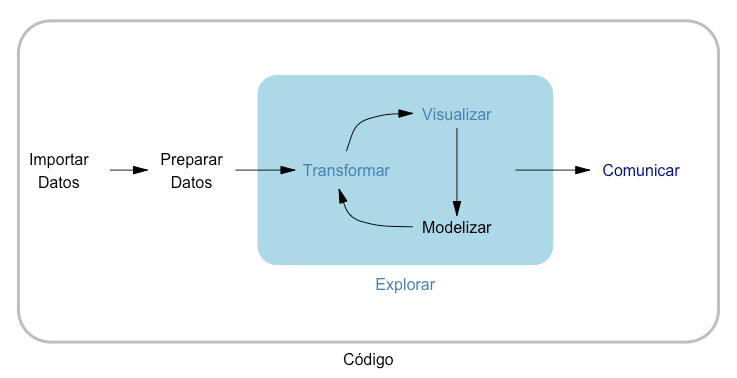
Figura 3.1: Flujo de trabajo en ciencia de datos propuesto por el \(tidyverse\). Fuente: Wickham and Grolemund (2016).
Además del mencionado libro, la web del tidyverse (http://tidyverse.org) contiene toda la documentación de los paquetes, incluidos artículos para tareas concretas, que merece la pena leer alguna vez. En la web están también las conocidas como cheatsheets, algunas de ellas disponibles también en la ayuda de RStudio (menú Help/Cheatsheets).
Dentro del flujo de trabajo de la Fig. 3.1, ya se ha tratado
la primera etapa (Import) en la Sec. 3.5.2. Es importante
señalar que, al utilizar las funciones del tidyverse, los datos se organizan
en objetos de clase tibble, que es una extensión del data.frame de R base.
Las principales diferencias son:
- Permite una representación compacta en la consola al mostrar la tabla de datos.
- La selección con corchetes simples de una única variable siempre devuelve
otro
tibble(a diferencia de undata.frame, que devuelve un vector).
Se puede forzar a que una tabla de datos sea de un tipo u otro con las funciones as.data.frame (de tibble a data.frame) y as_tibble (de data.frame a tibble).
Siguiendo con el esquema de la Fig. 3.1, en este apartado se verán algunas tareas de las etapas Tidy (organizar) y Transform (transformar), que serán ampliadas en los Cap. 8 y 9. La visualización (Visualise) se tratará específicamente en el Cap. 11 y transversalmente en muchos otros. La modelización (Model) se trata extensamente en los capítulos de las partes IV a IX, y la comunicación (Communicate) se verá en los capítulos de la Parte X. Una de las características de la forma en que están programados los paquetes del tidyverse es que se puede trabajar13 con pipes.
El pipe es, básicamente, un operador compuesto de dos caracteres, |>,
que se puede obtener con el atajo de teclado CTRL+MAYUS+M. El operador
se pone en medio de dos expresiones de R, sean lado_izquierdo
y lado_derecho las expresiones que se ponen a izquierda y derecha
del pipe. Entonces se utiliza de la siguiente manera:
Nota
El operador nativo de R, |>, apareció en la versión R-4.1.0. Hay un operador alternativo que proviene del paquete magrittr, %>%, que había que usar antes de esta versión, y mucha literatura y documentación está escrita usándolo. Hay diferencias, pero a los efectos de este capítulo ambos operadores se pueden utilizar indistintamente.
La expresión lado_izquierdo debe producir un valor, que puede
ser cualquier objeto de R.
La expresión lado_derecho debe ser una función, que tomará como primer
argumento el valor producido en la parte izquierda.
Si se desea guardar el resultado final, se debe asignar el resultado
a algún nombre de objeto para que se almacene en el espacio de trabajo.
La siguiente expresión sería un ejemplo de uso.
La ventaja de usar los pipes es que se pueden encadenar, de forma que el resultado de cada operación pasa a la siguiente expresión del pipeline (secuencia de operaciones con pipe), como en el siguiente ejemplo:
3.6.2 Transformación de datos con dplyr
En la gramática del tidyverse, dentro del paquete dplyr se dispone de una
serie de “verbos” (funciones) para una sola tabla, que se pueden
agrupar en tres categorías: para trabajar con filas, para trabajar con
columnas y para resumir datos.
3.6.2.1 Operaciones con filas
Los verbos definidos para estas operaciones son:
-
filter(): elige filas en función de los valores de la columna.
pm10 <- contam_mad |>
filter(nom_abv == "PM10") # se filtra por PM10-
arrange(): cambia el orden de las filas con algún criterio.
-
slice(): extrae filas por su índice. También hay una serie de funciones “asistentes” (helpers) para obtener los índices que se utilizan con frecuencia. Por ejemplo:-
slice_head()yslice_tail()obtienen las primeras y últimas filas respectivamente (por defecto, una). Se puede especificarn(número) oprop(proporción) de filas. -
slice_sample()obtiene una muestra aleatoria denfilas (o proporciónprop). -
slice_min(),slice_max()obtienen las filas que contienen los menores o mayores valores respectivamente de la variable indicada en el argumentoorder_by. Si no se especificanoprop, se obtienen solo las filas que contienen el mínimo o el máximo. Nótese que puede haber más de una fila que cumpla la condición.
-
Véase el resultado de los siguientes ejemplos:
pm10 |> slice(10:15) # extrae filas desde la 10 a la 15
pm10 |> slice_tail(n = 3) # extrae las tres últimas filas
pm10 |> slice_max(order_by = daily_mean) # día con mayor valor medio de PM10
set.seed(1) # Para que la muestra aleatoria sea reproducible
pm10 |> slice_sample(n = 4) # muestra 4 registros3.6.2.2 Operaciones con columnas
Los verbos definidos para estas operaciones son:
-
select(): indica cuando una columna se incluye o no. Se pueden utilizar helpers para seleccionar columnas que cumplan cierta condición (por ejemplo, ser numéricas) y también para “quitar” columnas de la selección (con el signo menos, [-]).
pm10 |> select(longitud, latitud, daily_mean, tipo)
pm10 |> select(where(is.numeric))
pm10 |> select(-c(id:latitud))En cuanto a la modificación de datos, existen múltiples posibilidades. Algunas de ellas son:
rename(): cambia el nombre de la columna.mutate(): cambia los valores de las columnas y crea nuevas columnas. La funcióntransmute()funciona igual quemutate(), pero la tabla de datos resultante solo contiene las nuevas columnas creadas.relocate(): cambia el orden de las columnas.
pm10 |> rename(zona_calidad_aire = zona)
pm10 |> relocate(fecha, .before = estaciones)
pm10_na <- pm10 |> mutate(isna = is.na(daily_mean))En este punto, es importante señalar que dentro de la función mutate()
se puede usar cualquier función vectorizada para transformar las
variables. Por ejemplo, se podría transformar una columna con las funciones
as.xxx que se vieron en la Sec. 3.5.1, aplicar formatos
a fechas o usar funciones del paquete lubridate para trabajar con este
tipo de datos. A medida que se avance en el libro irán apareciendo aplicaciones
que ahora, quizás, no sean tan evidentes.
3.6.2.3 Operaciones de resumen y agrupación
La primera operación de resumen que puede surgir es
“contar” filas. La función tally() devuelve el número de filas totales
de un data.frame. La función count() proporciona también este número; si, además, se pasa como argumento alguna variable, lo que devuelve es el número
de filas para cada valor diferente de dicha/s variable/s. Estos recuentos se
pueden añadir a la tabla de datos con las funciones add_count() y add_tally(),
lo que permite calcular frecuencias absolutas y relativas fácilmente.
pm10 |> tally()
#> n
#> 1 53794
pm10 |> count(zona)
#> zona n
#> 1: Interior M30 20690
#> 2: Noreste 12414
#> 3: Noroeste 4138
#> 4: Sureste 8276
#> 5: Suroeste 8276La función summarise() (o, equivalentemente, summarize()) aplica alguna
función de resumen a la/s variable/s que se especifiquen (mean(), max(), etc.).
El paquete dplyr tiene algunas funciones de resumen adicionales, como
n() (número de filas), n_distinct() (número de filas con valores distintos) y
first(), last(), nth() (primero, último y n-ésimo valor, en el orden en el que se encuentran, respectivamente).
En muchas ocasiones, las operaciones de análisis se realizan en grupos
definidos por alguna variable de agrupación. La función group_by()
“prepara” la tabla de datos para realizar operaciones de este tipo.
Una vez agrupados los datos, se pueden añadir operaciones de
resumen como las vistas anteriormente. A veces hay que “desagrupar” los
datos, para lo que se utiliza la función ungroup().
A continuación, se muestra una expresión un poco más compleja que las anteriores.
En el conjunto de datos contam_mad del paquete CDR, se filtra por el
nombre de contaminante “NOx”. Después se agrupan los datos por zona y se
calculan algunos estadísticos de resumen para cada zona.
contam_mad |>
filter(nom_abv == "NOx") |> # se filtra por N0x
group_by(zona) |>
summarize(
min = min(daily_mean, na.rm = TRUE),
q1 = quantile(daily_mean, 0.25, na.rm = TRUE),
median = median(daily_mean, na.rm = TRUE),
mean = mean(daily_mean, na.rm = TRUE),
q3 = quantile(daily_mean, 0.75, na.rm = TRUE),
max = max(daily_mean, na.rm = TRUE)
)
#> A tibble: 5 × 7
zona min q1 median mean q3 max
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Interior M30 0.0833 32.4 54.1 72.9 90.0 759.
#> 2 Noreste 1 23.8 39.6 56.2 68.9 516.
#> 3 Noroeste 0 12.0 20.3 29.7 34.5 352.
#> 4 Sureste 0 29.1 45.4 64.6 77.2 453
#> 5 Suroeste 0.667 33.5 59.6 90.5 114. 666.3.6.3 Combinación de datos
En el apartado anterior se han tratado los “verbos” de una tabla. Es muy común que haya que combinar datos de distintas tablas, para lo cual se utilizan lo que el tidyverse considera two tables verbs. En esencia, para combinar tablas que contienen información relacionada, hay que saber cuáles son las columnas que se refieren a lo mismo, para hacer las uniones (joins) utilizando esas columnas. Hay cuatro tipos de uniones que se pueden realizar, usando las siguientes funciones:
-
inner_join(): se incluyen las filas de ambas tablas para las que coinciden las variables de unión. -
left_join(): se incluyen todas las filas de la primera tabla y solo las de la segunda donde hay coincidencias. -
right_join(): se incluyen todas las filas de la segunda tabla y solo las de la primera donde hay coincidencias. -
full_join(): se incluyen todas las filas de las dos tablas.
Las funciones requieren como argumentos dos tablas de datos y
la especificación de las columnas coincidentes. Si no se especifica,
hace las uniones por todas las columnas coincidentes en ambas tablas. Para
las filas que solo están en una de las tablas, se añaden valores NA
donde no haya coincidencias.
A modo de ejemplo, las siguientes expresiones unen dos datasets para combinar datos de municipios con su renta. En el Cap. 8 se verán estas uniones en la práctica.
library("sf")
munis_renta <- municipios |>
left_join(renta_municipio_data) |>
select(name, cpro, cmun, `2019`)
#> Joining, by = "codigo_ine"Otra forma de unir tablas es, simplemente, añadiendo columnas (que tengan el mismo
número de filas) o filas (que tengan el mismo número de columnas). Para ello se usan
las funciones bind_cols() y bind_rows(), respectivamente.
Otra forma conveniente de añadir nuevas filas o columnas son las funciones
add_row() y add_column(). Se pueden añadir antes o después de una fila/columna
especificada con el argumento .before, y pasando los valores como pares “variable = valor” para cada variable en el conjunto de datos.
Como comentario final del paquete dplyr, una característica importante es que
se pueden usar las funciones vistas sobre
tablas de una base de datos, sin necesidad de utilizar sentencias SQL y con
la ventaja de que las operaciones se realizan en el motor de la base de datos.
En el Cap. 5 se tratarán las cuestiones relacionadas con los gestores de bases de datos y SQL.
3.6.4 Reorganización de datos
A lo largo del capítulo se ha visto la importancia de disponer los datos de forma rectangular, de forma que se tenga una columna para cada variable y una fila para cada observación. Algunas veces es conveniente reorganizar los datos más “a lo ancho” o más “a lo largo” de lo que se encuentran.
Para estas operaciones se utilizan las funciones pivot_longer() y
pivot_wider() del paquete tidyr del tidyverse de la siguiente forma:
pivot_longer(): el argumentonames_toasigna el nombre de la nueva variable que va a indicar de qué columna vienen los datos; y el argumentovalues_toasigna el nombre de la nueva variable que va a contener el valor de la tabla original.pivot_wider(): el argumentonames_fromindica el nombre de la variable que contiene los nombres de las nuevas columnas a crear a lo ancho; y el argumentovalues_fromindica el nombre de la variable que contiene los valores en la tabla original. Las observaciones deben estar identificadas de forma única por varias variables. Si no es el caso, se puede aplicar una función al estilo de las tablas dinámicas de las hojas de cálculo con el argumentovalues_fn.
Nota
Las funciones pivot_longer() y pivot_wider() admiten otros argumentos
names_xx y values_xx para personalizar la forma de reestructurar los datos.
En la mayoría de las
ocasiones será suficiente con las comentadas (xx_from y xx_to). Si fuera necesario, se recomienda consultar la ayuda
de las funciones, o la lectura del artículo sobre pivoting.
A modo de ejemplo, el conjunto de datos contam_mad tiene los datos “mezclados” de varias variables medioambientales en la columna daily_mean. La columna nom_abv contiene el parámetro al que se refiere la columna de datos. Entonces, interesa “extender” la tabla para tener cada parámetro en una columna, de forma que se pueda hacer un análisis de datos adecuado, como en el siguiente código:
library("tidyr")
extendida <- contam_mad |>
pivot_wider(names_from = "nom_abv",
values_from = "daily_mean",
values_fn = mean)
colnames(extendida)
#> [1] "estaciones" "id" "id_name" "longitud"
#> [5] "latitud" "nom_mag" "ud_med" "fecha"
#> [9] "zona" "tipo" "BEN" "SO2"
#> [13] "NO2" "EBE" "CO" "NO"
#> [17] "PM10" "PM2.5" "TOL" "NOx"Se deja como ejercicio volver a obtener la tabla original usando la función pivot_longer() a partir del objeto extendida.
El paquete tidyr también contiene funciones para reorganizar las columnas
de la tabla uniendo columnas con la función unite(), o separando una columna
en dos o más con la función separate() (véanse los detalles en la ayuda de las funciones).
Para terminar este apartado de reorganización de datos, se da una
primera aproximación al tratamiento de valores perdidos, que se abordará en el
Cap. 8. En R, un valor
perdido se representa por el valor especial NA (not available). Brevemente,
las funciones más utilizadas en este campo son:
drop_na()del paquetetidyr: permite eliminar las filas que tienen valores perdidos en ciertas variables (o en cualquiera, si no se especifica ninguna).replace_na(): sustituye los valores perdidos en cada variable por el valor especificado.fill(): permite “rellenar” valores perdidos con los últimos encontrados.
Los datos de contaminación a menudo tienen muchos valores perdidos. La siguiente expresión elimina las filas del conjunto de datos contam_mad con valores perdidos y, después, cuenta las filas.
Resumen
R es software libre y gratuito, mantenido por una enorme comunidad.
La forma de interactuar con R es mediante expresiones, que se escriben en scripts, y al ejecutarlas se obtienen los resultados.
Los objetos de datos que se vayan a usar deben estar en el espacio de trabajo.
RStudio es un “envoltorio” de R, y por tanto R tiene que estar instalado en el sistema para poder usar RStudio.
Los paquetes se instalan una sola vez, y deben cargarse con
library()para usar sus funciones.La tabla de datos o
data.framees la estructura de datos más adecuada para análisis de datos y cada columna es unvector.El tidyverse es un conjunto de paquetes que facilita las tareas de análisis de datos.
El operador pipe,
|>, permite “pasar” valores a funciones de forma encadenada.Las operaciones básicas con una tabla son filtrado, selección y resumen.
Para crear nuevas columnas en las tablas de datos se usa la función
mutate.Para combinar tablas con columnas comunes se usan las funciones
xx_join.